2月27日に、姉が勤める理化学研究所(情報基盤センター)を見学してきました。ずっと行きたいなと思っていたのですが、なかなか機会がなく、今回の休暇を利用してようやく実現しました。
入り口でパスをもらい、早速、リニューアルしたばかりという4Dビジュアライゼーションシアターから見学スタートです。
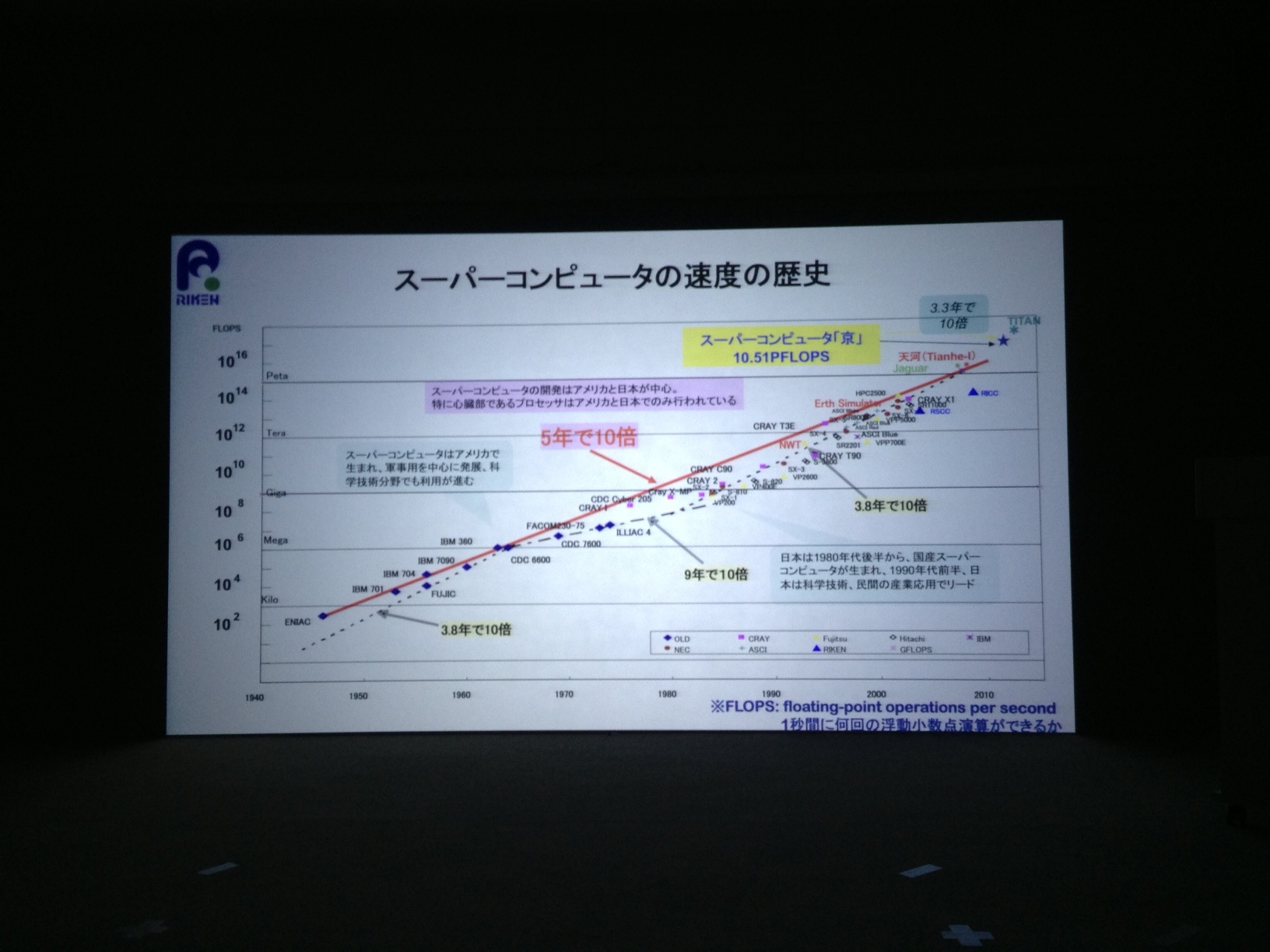
まずは、スーパーコンピュータの歴史など、基本的なスライドをさらっと見せてもらう。きちんと見学に来た方たちを対応するときは、ここももう少しじっくり説明されるのかもしれませんが、私はとりあえずスライドをざざっと見せてもらう形で駆け足で。
速度の歴史。5年で10倍。
ちょっと部屋が暗くて写真がキレイに撮れず、上の写真を含め、以降、もやっとした写真が続いてしまうのですが、シャキっと撮れている写真がほとんどありませんでした......。ブログ書きだすとカメラ欲しくなるね。
次に、いくつかのデモを見せてもらい、計算機を使ってどんな研究がされているのか、何に役立てられているのかを教えてもらいました。
最初は、車の周りで起きる空気の流れのシミュレーション。これだけ動画を撮っていました。
ちょっとぼんやりなのは3Dで見ていたのをそのまま撮っていたからかな......。
次に、ボールを投げたときの空気の流れのシミュレーション。ここからは写真。
これらは「空気力学」というもので、一つひとつの空気の流れがどういう影響を与えるかについても説明してもらったのですが、長くなるので割愛。とにかく、空気の作用、空気中を運動する物に与える影響などがわかるということですね。
次、ザリガニ。
本物のザリガニをCTみたいなものでデータをとって再現しているそうです。
なんでそんなことをするのか? というと、機械とか工業製品とかは設計図があって、そのデータをもとにシミュレーション(例えば自動車の衝突とか)ができるけど、人間の体とか生き物、植物には設計図が無いから、その形を正確に作りたいときは、CTとか、何かしらデータをとって作るしかないから。ただ、こういうデータは計算機で計算するのに大変向いておらず、それを計算機で扱いやすいようにするための研究プロジェクトみたいなのがあったそうで、このザリガニのシミュレーションはその一環とのこと。
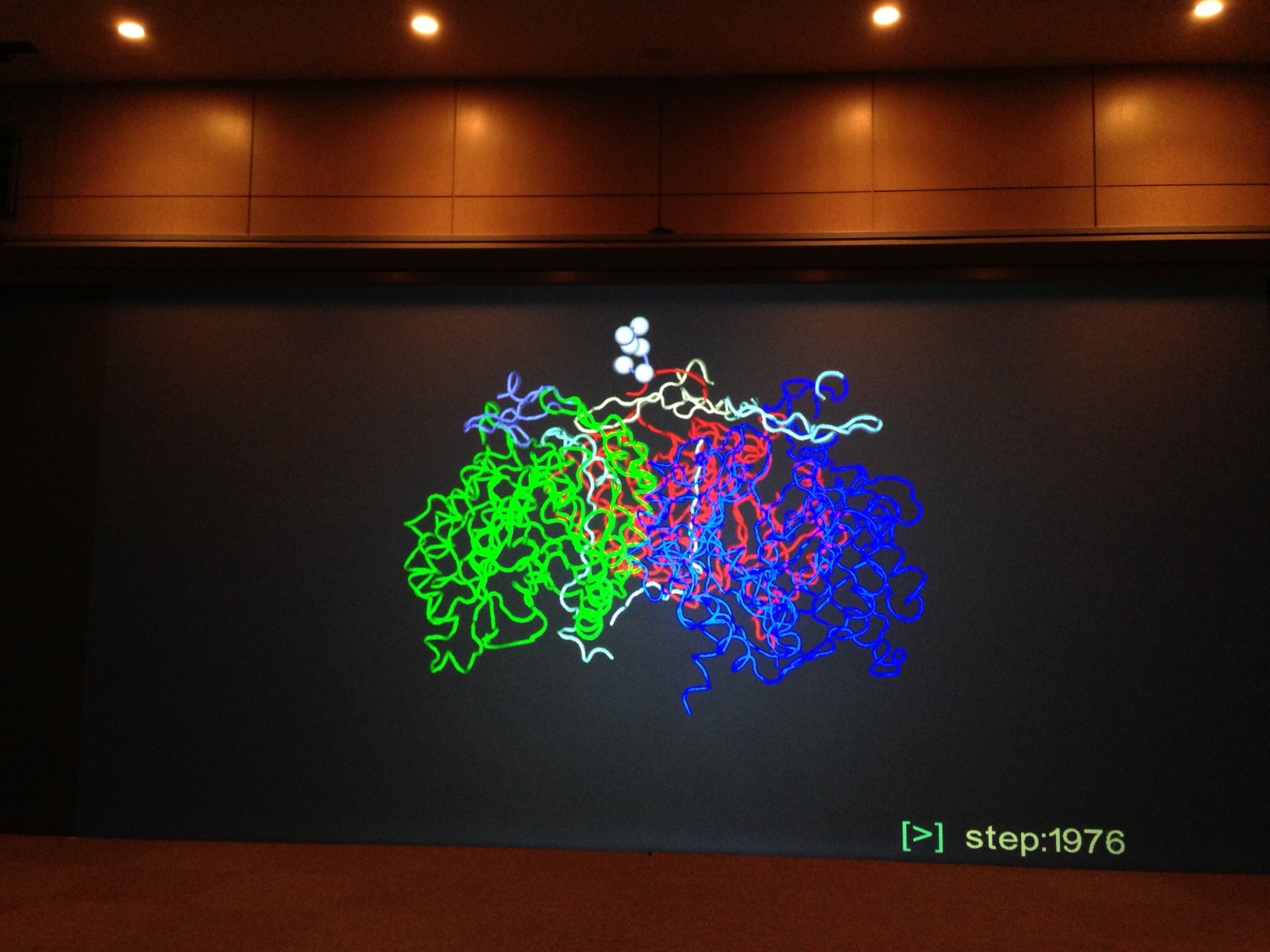
最後に、タンパク質が悪さをして抗生物質を細胞の外に出しちゃうというシミュレーション。
「抗生物質が効かなくなる」という症状を聞いたことがありますが、そのメカニズムを「分子動力学」というのを用いて検証した、ということだそうです。
一般人に(というか私に)わかるように簡単な言葉で超ざっくり説明してもらったところ......。
- 分子っていうのは常に動いている(ブラウン運動)。
- とはいえ別に分子に意思を持って動いているわけではない。
- でもその構造とか、電気的な「引き合う」とか「離れる」などの相互作用で、「形」があるがゆえに、何か意味のある動きになることがある。
- つまり、こんな構造で、温度がこれぐらいで......といった、ある条件の下では、結果的にこのタンパク質がこう動いて、抗生物質を外に出しちゃうよね。
というのを検証したシミュレーション、とのこと。結果そうなってしまう、ということなんだけど、それは生命現象の根幹なのだとか。
本当にざっくり教えてもらっているだけなので、こんな説明ですみません。一応説明してもらったとおり書いているつもりなのですが、「ちゃうわ! 何聞いとんねん!」だったら連絡ください(->姉)。
実際見せてもらっているものはもちろんスクリーン上で動いており、3Dで見ているのですが、写真撮影用に2Dでしてもらって撮りました。
これらは、Linuxのクラスターで動いているんだよ、とのことで、それも見せてもらいました。
スクリーンの裏側に。まあここは地味な感じ。
iPadでも操作できるようにしたそうです。操作しているイシダイみたいなのが姉。
最後に計算機室(?)も見せてもらいました。
空冷式。表から吸って裏から出す。吸わせてる側が cold aisle、吐き出してる側が hot aisle。なるほど。
これってWEBとかのサーバーと何が違うの? というと、まあいろいろあるけれど、とにかく「インターコネクトが速い」のだそうです。すべてのノードが「ツリー構造」で物理的に接続されているのですって。ほほう。
ちなみに、「京」の場合は、ここで使っている計算機の100倍近い性能(64万コア......と言われたけれど、正直よくわかっていない・笑)があって、それを効率よくつなごうとすると、ツリー構造では無理なので「3Dトーラス」という形で接続されている......と。ほほう。もう聞いたことない(笑)。で、そうなると、スケジューリングの仕方なども変える必要があり、そこがすごく難しくてお金がかかるそうです。
これは MDGRAPE-3。
GRAPE といえば、『スーパーコンピューターを20万円で創る』を読んだことがあり、国立天文台の小久保英一郎先生を取材させてもらったことがある(※)私としては、聞きなじみのある名前です。
→『スーパーコンピューターを20万円で創る』(伊藤智義著・集英社刊)
※小久保先生に取材したのは英語についてのお話で、研究は関係ありません(^_^;)
→ 理論天文学者小久保英一郎先生に聞く"理系的"英語学習法
天文シミュレーション用に開発された計算機がGRAPEですが、その後、理研がこれをベースに分子動力学専用のMDGRAPEを開発したのだそうです。MD = Molecular Dynamics ですね。
MDGRAPEは単体で動いているのではなく、PCIでCPUにつながっていて、それが下の写真の SGI の Altix XE というPCクラスタ。
写真がとても下手くそで申し訳ないのですが、かっこいいねえ。
ひと通り見学させてもらい、所内にあるタリーズで休憩。帰りは夕日がとてもきれいでした。

そして私はもう少しちゃんと理解したいので、これを読んでみたいとおもいます。